在地球的生态系统中,微生物作为最为丰富且多样的生命形式,深刻影响着地球的物质循环、生态平衡乃至人类健康。然而,自然环境中超过 99% 的微生物无法通过传统实验室培养方法获得纯培养,这一 “培养瓶颈” 长期制约着人类对微生物世界的认知。环境基因组学(Environmental Genomics)的兴起与发展,为突破这一困境提供了革命性的研究手段。作为宏基因组学的核心分支,环境基因组学通过直接获取环境样品中的全部遗传物质并进行系统分析,无需依赖微生物的分离培养,便能全面揭示微生物群落的物种组成、基因多样性及其生态功能。近 20 年来,随着第二代高通量测序技术(NGS)的大规模应用和 DNA 测序成本的显著降低(降幅超过 10⁵倍),环境基因组学已迅速发展成为生物学与环境科学交叉领域的重要研究方向,正以前所未有的方式重塑传统环境生物学与生态学的研究范式。
一、环境基因组学的技术框架与核心优势
环境基因组学的研究对象是环境样品中微生物群落的全部基因组总和,其技术流程涵盖从样品采集、核酸提取到测序分析、数据解读的完整链条。与传统微生物研究方法相比,环境基因组学具有显著优势:它无需对微生物进行富集培养或核酸扩增,有效规避了培养依赖性、引物设计偏差和扩增偏好性等问题,能够更真实地反映微生物群落在原位环境中的生物多样性及组成丰度信息。这种 “无偏性” 研究思路使其成为探索未知微生物世界的关键工具,广义上涵盖了环境基因组、生态基因组学和群体基因组学的研究目标与内容,因此也被称为 “泛宏基因组”(Bulk metagenome)研究。随着分子生物学技术的自动化与商业化发展,环境基因组学研究已从早期低通量、高劳动密集型的 “湿实验”,转变为高通量、数据密集型的 “干实验”,生物信息学与生物统计学的深度应用成为推动领域发展的核心动力。
二、新微生物物种的发现:解锁 “微生物暗物质”
环境基因组学的突破性进展,首先体现在对新微生物物种的持续发现上。自然环境中大量未培养微生物被称为 “微生物暗物质”,而环境基因组学通过宏基因组组装基因组(Metagenome-Assembled Genome, MAG)技术,为解锁这些暗物质提供了可能。美国加州大学伯克利分校 Jill Banfield 教授团队是该领域的先驱,他们通过对科罗拉多河附近蓄水层微生物组的 MAG 分析与转录组研究,发现了一类被命名为 “候选门辐射群”(Candidate Phyla Radiation, CPR)的特殊微生物。这类微生物颠覆了传统原核生物认知:其基因组中存在外显子与内含子,核糖体 RNA 蛋白质编码基因中还含有自我剪接的内含子。该成果于 2015 年发表在《自然》杂志后,引发了学界对未培养微生物的广泛关注。此外,全球公共数据库中积累的海量宏基因组数据为新物种发现提供了另一重要途径。美国能源部联合基因研究所(JGI)团队通过对 JGI-IMG/M 数据库中 5.2 TB 宏基因组数据的深度挖掘,于 2016 年发现了新候选细菌门 Candidatus Kryptonia,这类异养微生物主要栖息于高温、中性 pH 的地热泉环境中,成为通过公共数据重分析发现新物种的典型案例,充分体现了环境基因组学研究微生物暗物质解锁的核心价值。
三、新酶基因与功能基因簇的挖掘:从基础研究到应用转化
在新酶基因与功能基因簇的挖掘方面,环境基因组学展现出巨大潜力。CRISPR-Cas 系统的发现与应用是最具代表性的成果之一。Jill Banfield 教授与 Jennifer Doudna 教授团队合作,对地下水、土壤、婴儿肠道等环境样品的 TB 级宏基因组数据进行解析,通过组装与分箱技术获得 1.55 亿个蛋白编码基因及大量 MAGs,最终发现了首个来自古菌域的 Cas9 蛋白,以及两种新型 CRISPR-Cas 系统(CRISPR-CasX 和 CRISPR-CasY)。这一发现为基因编辑技术提供了新工具,而 Doudna 教授也因在 CRISPR-Cas 系统领域的贡献获得 2020 年诺贝尔化学奖。环境基因组学在新型抗生素研发中同样发挥关键作用。传统抗生素多来自可培养的放线菌、芽孢杆菌等少数微生物类群,而环境基因组学帮助研究者从土壤未培养微生物中发现了大量新型生物合成基因簇。美国洛克菲勒大学团队通过宏基因组筛选,从 2000 份土壤样品中发现了一个未知抗生素合成基因簇,并成功合成新型钙依赖环肽抗生素 “马拉西啶”(Malacidins),该抗生素可有效杀灭多重耐药革兰氏阳性致病菌,为解决抗生素耐药性问题提供了新希望,展现了环境基因组学研究功能基因簇挖掘应用的巨大潜力。
四、代谢途径的全新认知:重塑微生物生态学理论
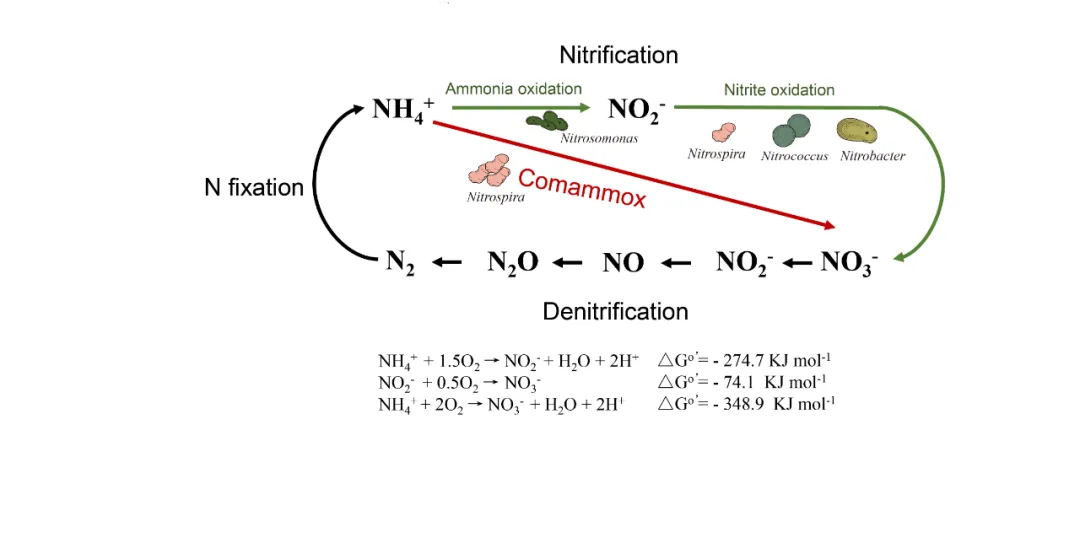
环境基因组学还推动了对微生物代谢途径的全新认知,彻底改写了传统微生物生态学理论。完全氨氧化(Comammox)微生物的发现便是典型例证。2015 年,奥地利 Wagner Michael 教授与荷兰 Sebastian Lücker 教授团队在《自然》杂志同期发表论文,通过宏基因组测序、同位素示踪与 MAG 分析,证实了完全氨氧化 Nitrospira 细菌能够独立将氨氮直接氧化为硝酸盐。这一发现破解了困扰学界百年的谜题,打破了 “硝化作用必须由氨氧化菌与亚硝酸盐氧化菌分两步完成” 的传统认知,为氮循环研究提供了新视角。类似地,通过环境基因组学技术,研究者还发现了深古菌门参与的硝酸亚还原厌氧甲烷化等新型代谢过程,揭示了未培养微生物在全球生物地球化学循环中的关键作用。这些研究表明,将环境基因组学与生物反应器富集、稳定同位素示踪等技术结合,是揭示微生物功能与代谢潜力的有效手段,深化了环境基因组学研究微生物代谢途径解析的理论认知。
五、微生物组 - 环境 - 效能关联:从理论到应用的桥梁
在探索微生物组与环境及系统效能的关联方面,环境基因组学为理论研究向实际应用转化搭建了桥梁。通过解析微生物群落结构与功能,研究者可定向调控微生物组以服务于农业、环境修复与资源回收等领域。例如,通过优化土壤微生物群落提高作物生产力,通过微生物菌剂修复污染水体,或通过富集功能微生物实现废水资源回收等。香港大学研究团队通过 16S rRNA 扩增子测序与多参数统计分析,揭示了城镇污水处理厂中微生物组(M)、环境参数(E)与系统效能(P)的内在关联,提出了基于微生物组调控的活性污泥起泡问题解决方案。美国科研团队提出的 “设计 - 建造 - 测试 - 学习(DBTL)” 体系,更是将环境基因组学作为核心工具,为微生物组在医药、能源等领域的应用提供了系统性理论指导。
六、挑战与未来展望
尽管环境基因组学已取得显著成就,但对未培养微生物的认知仍存在巨大空白。样品采集、DNA 提取与测序过程中的偏差可能影响结果准确性,因此标准化操作流程的制定与优化至关重要。此外,基因组水平的功能预测不等于实际活性,如何实现微生物组成与功能的耦合研究仍是核心挑战。当前,稳定性同位素示踪、转录组学、代谢组学等技术与环境基因组学的结合,为解决这一问题提供了新思路。未来,随着技术方法的不断创新与数据积累,环境基因组学必将在揭示生命奥秘、推动生物技术革新与服务人类社会发展中发挥更加重要的作用,持续为微生物世界的探索带来新的惊喜与突破。
乐备实是国内专注于提供高质量蛋白检测以及组学分析服务的实验服务专家,自2018年成立以来,乐备实不断寻求突破,公司的服务技术平台已扩展到单细胞测序、空间多组学、流式检测、超敏电化学发光、Luminex多因子检测、抗体芯片、PCR Array、ELISA、Elispot、多色免疫组化等30多个,建立起了一套涵盖基因、蛋白、细胞以及组织水平实验的完整检测体系。
我们可提供从样本运输、储存管理、样本制备、样本检测到检测数据分析的全流程服务。凭借严格的实验室管理流程、标准化实验室操作、原始数据储存体系以及实验项目管理系统,已经为超过3000家客户单位提供服务,年检测样本超过100万,受到了广大客户的信任与支持。











 沪公网安备31011502400759号
沪公网安备31011502400759号
 营业执照(三证合一)
营业执照(三证合一)


