一、染色质重塑复合物识别与滑动六聚体核小体的结构机制解析
在真核细胞中,基因组DNA缠绕于组蛋白八聚体形成核小体,此结构是维持染色质高级构象及调控基因表达的核心基础。经典核小体由约147 bp的DNA片段与一个组蛋白八聚体(包含两个H2A-H2B二聚体及一个H3-H4四聚体)组装而成。然而,在动态的细胞环境中,核小体组成并非恒定。诸如DNA复制、转录及染色质重塑等多种生物学过程可导致其组蛋白组成与DNA包裹方式发生改变,从而形成各类非经典核小体。
其中,六聚体核小体是研究较为深入的非经典形式之一。其结构仅包含一个H2A-H2B二聚体与一个H3-H4四聚体,所结合的DNA长度相应缩短至约110 bp。该结构早期即被发现与转录活性相关:1983年,Daniela Rhodes团队的研究表明六聚体核小体可与RNA聚合酶II结合,提示其在活跃转录染色质区域存在。2022年,Geeta Narlikar实验室进一步发现,六聚体核小体可作为底物被酵母源的染色质重塑复合物INO80所识别并驱动其滑动,这引发了领域内对其特异性识别与重塑机制的深入关注。
2023年6月29日,欧洲分子生物学实验室Sebastian Eustermann课题组的张敏博士等在《Science》上发表了题为“Hexasome-INO80 complex reveals structural basis of non-canonical nucleosome remodeling”的研究论文。该研究首次解析了染色质重塑复合物INO80与六聚体核小体形成功能复合物的高分辨率冷冻电镜结构,从根本上揭示了INO80复合物特异性识别、结合并驱动此类非经典核小体进行空间重排的分子机理。这一结构生物学突破,为理解六聚体核小体在基因组内参与转录调控、染色质可塑性维持及相关细胞过程的分子基础提供了关键框架。

二、INO80复合物与六聚体核小体结合的结构特征
INO80复合物通常由15个亚基组成,按其功能可划分为三个主要模块:含有Ino80 ATP水解酶的催化核心模块(core module)、包含核内肌动蛋白的Arp8模块(Arp8 module),以及物种特异的Nhp10模块(Nhp10 module)。其中,Nhp10模块对于复合物的核小体滑动活性并非必需(图1)。本研究采用重组表达技术,获得了嗜热毛壳菌(Chaetomium thermophilum)来源的INO80催化核心模块与Arp8模块,并在非交联条件下将其与六聚体核小体组装成复合物。通过单颗粒冷冻电镜技术,成功解析了该复合物在活性状态下的两种构象,其局部分辨率介于2.9至5.7 Å之间。
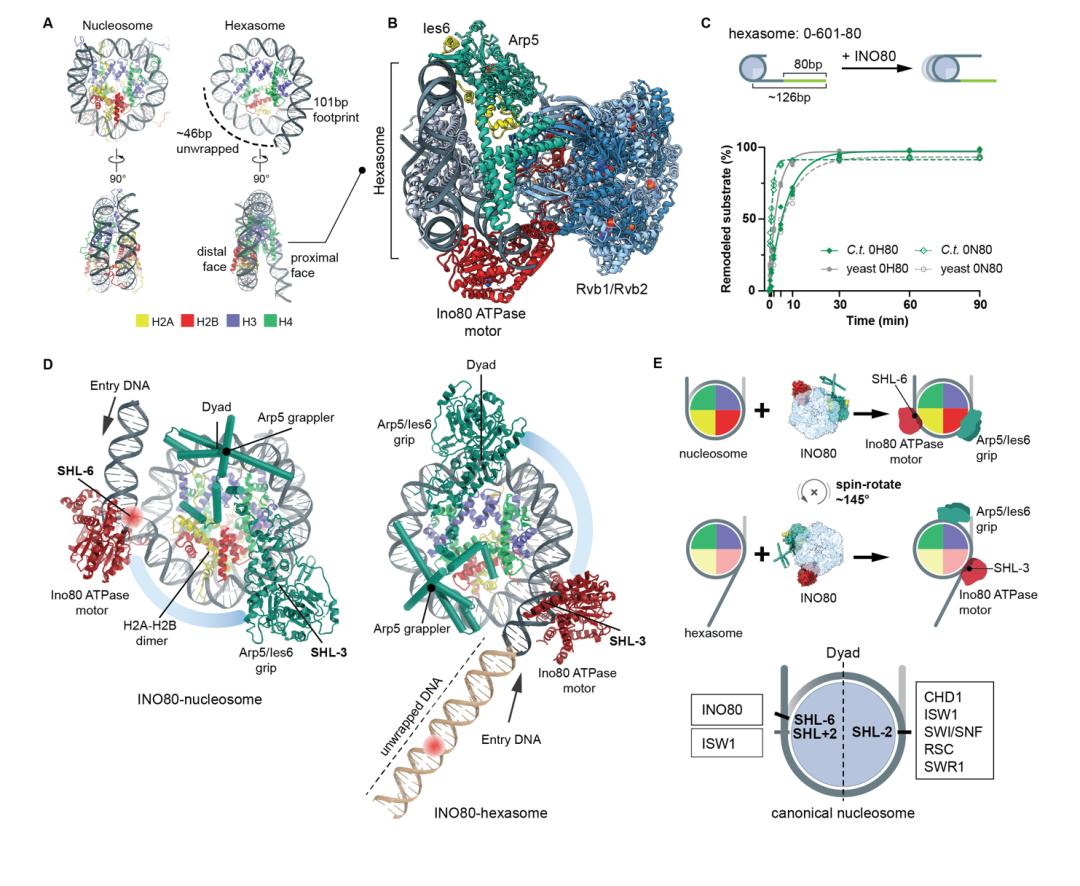 图1 INO80催化核心模块与六聚体核小体的总体结构
图1 INO80催化核心模块与六聚体核小体的总体结构
结构分析表明,INO80的催化核心模块主要与六聚体核小体的核心区域(即组蛋白六聚体及其所缠绕的DNA)结合(图1),而Arp8模块则特异性地结合于核小体间裸露的连接DNA(linker DNA)(图3,详见下文)。与此前解析的INO80与经典核小体的结合结构相比【5, 6】,其催化核心模块的自身构象未见显著改变;然而,该模块与六聚体核小体的结合模式却展现出根本性差异。INO80并非通过传统的酸性补丁(acidic patch)介导相互作用,而是选择性地结合在六聚体核小体上因缺失一个H2A-H2B二聚体而暴露出的蛋白表面。与此同时,六聚体核小体中仅存的单一H2A-H2B二聚体则朝向相反方向,完全暴露于溶剂之中(图1)。这一颠覆性的结构发现,后续也得到了生化实验的验证支持(图2)。
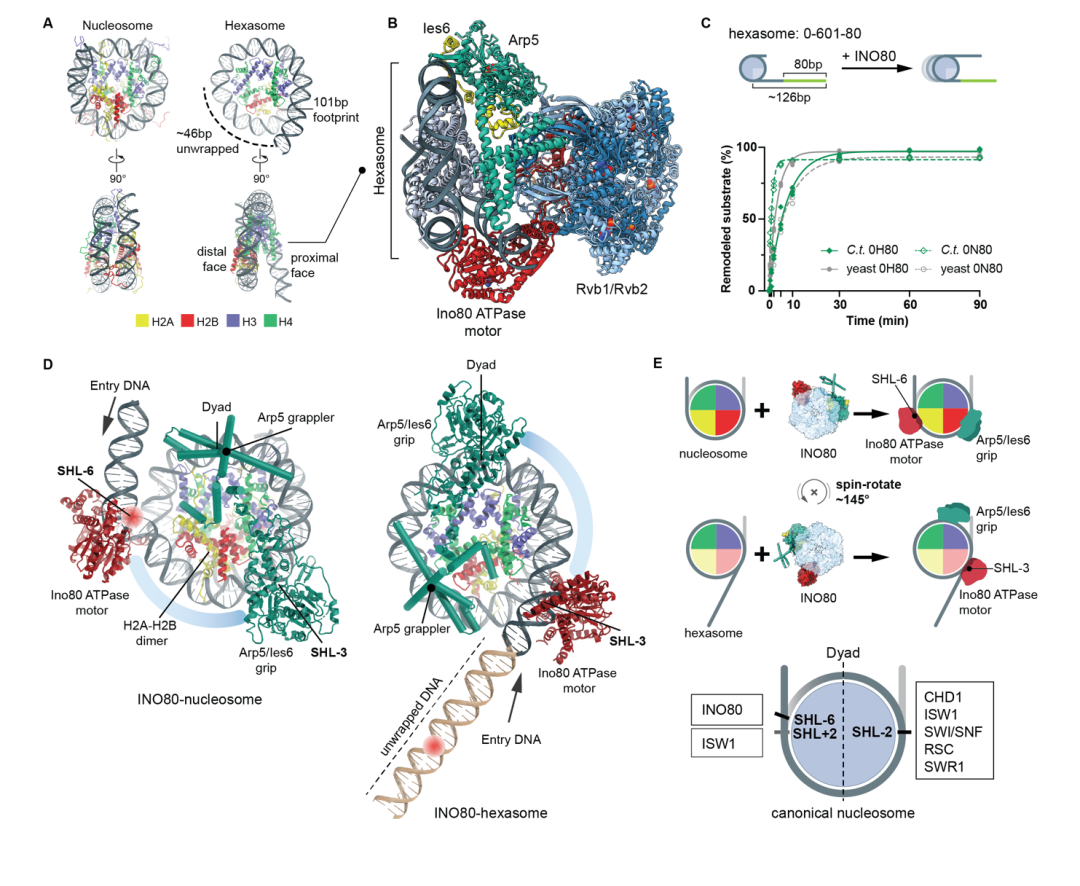 图2 非酸性补丁依赖的INO80-六聚体核小体识别机制
图2 非酸性补丁依赖的INO80-六聚体核小体识别机制
此外,相较于其在经典核小体上的定位,INO80复合物整体发生了约145°的中心旋转(spin-rotation)。这一显著的构象重排导致其马达亚基(Ino80 ATPase motor)结合位点从经典核小体上的SHL-6位置,转移至六聚体核小体DNA的SHL-3位置。值得注意的是,经典的SHL-6位点由于对应区域H2A-H2B二聚体的缺失,在六聚体核小体上已转变为一段非缠绕DNA(unwrapped DNA)区域(图1)。这些结构细节共同揭示了INO80复合物特异性识别并适应非经典核小体底物的独特分子机制。
已有研究指出,INO80复合物对经典核小体的滑动活性,高度依赖于其Arp5亚基与组蛋白H2A-H2B上酸性补丁(acidic patch)的相互作用【4,6】。在经典核小体结构中,Arp5亚基通过其“heel”和“foot”亚结构域直接结合至酸性补丁的酸性残基;针对这两个结构域的关键残基进行突变后,INO80的核小体滑动活性显著降低(图2)。然而,在六聚体核小体结构中,伴随一个H2A-H2B二聚体的缺失以及INO80整体发生约145°的中心旋转(spin-rotation),Arp5的“heel”与“foot”亚结构域转而与组蛋白H3-H4的表面区域接触。这一结构重塑解释了INO80在六聚体核小体上的滑动为何不依赖于经典的酸性补丁机制。
值得注意的是,与在经典核小体上的表现相反,Arp5“heel”和“foot”突变体在六聚体核小体上的滑动活性相较于野生型INO80反而呈现增强趋势。这一对比性发现表明,Arp5亚基在INO80复合物对核小体与六聚体核小体的滑动过程中,可能发挥着截然不同的调控作用。
三、Arp8模块作为连接DNA感应器的结构证据
由于INO80催化核心模块与Arp8模块之间存在显著的构象柔性,难以在单颗粒重构中将其作为整体进行处理。因此,本研究对Arp8模块进行了独立的颗粒挑取与三维重构分析。重构结果明确显示,Arp8模块直接结合于一段长约35 bp的裸露DNA片段。
为进一步解析两个模块之间的空间组织关系,研究者对参与上述重构的全部颗粒进行了单颗粒水平上的距离分布统计。分析结果表明,催化核心模块与Arp8模块平均相距约170 Å,并由一段长度约20 bp的DNA片段相连接(图3)。值得注意的是,通过对这些相距约170 Å的颗粒对进行整体重提取和二维分类,获得了同时包含催化核心模块与Arp8模块的二维类别图像(图3)。这一结果直接证实,在六聚体核小体复合物中,Arp8模块仍保持其作为“连接DNA感应器”的分子功能,通过识别并结合核小体间的连接DNA,参与调控复合物的结构与活性。
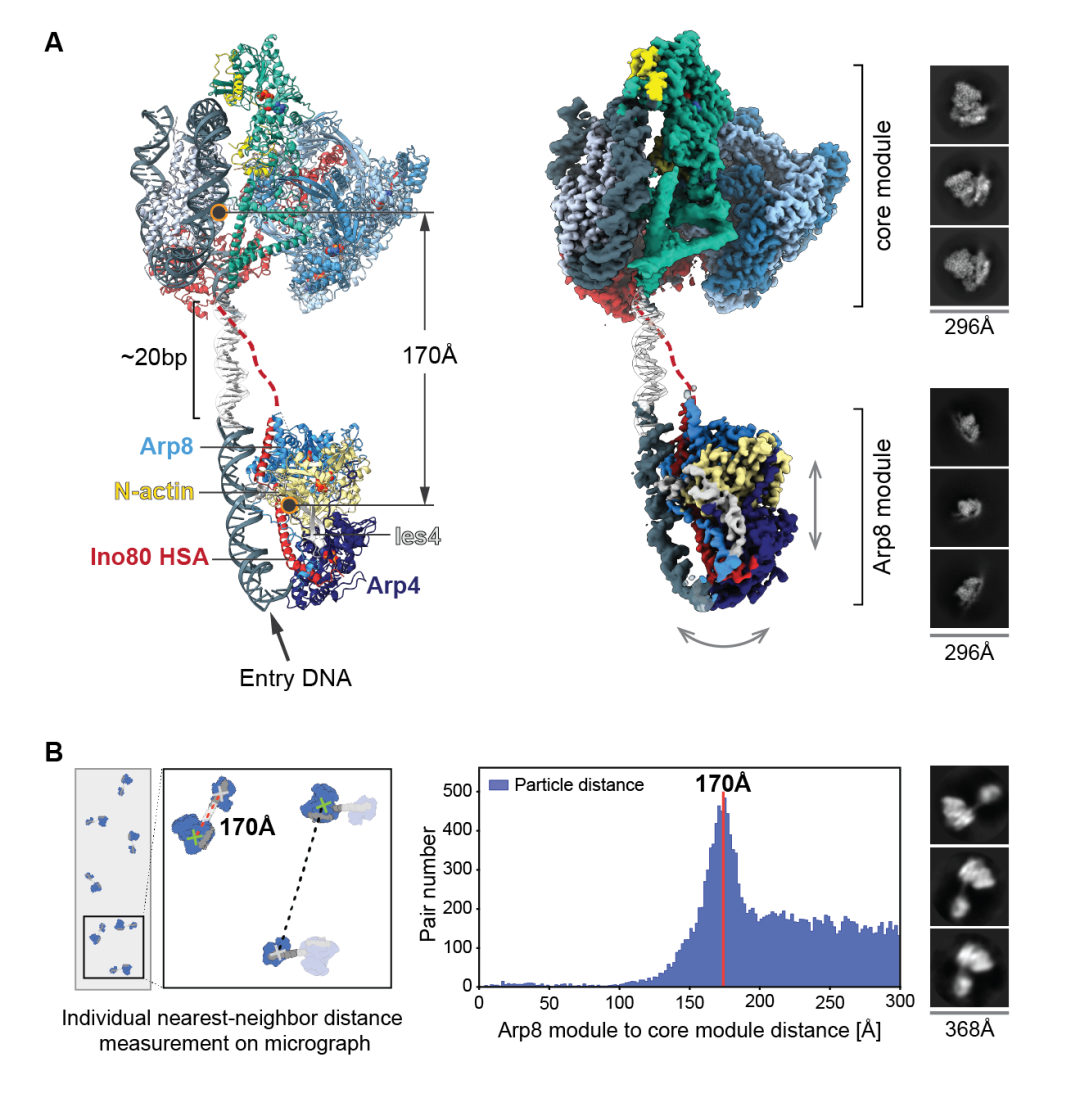 图3 Arp8模块与连接DNA的结构及其与催化核心模块的空间联系
图3 Arp8模块与连接DNA的结构及其与催化核心模块的空间联系
四、INO80的底物识别机制及其在染色质动力学中的功能意义
综上所述,INO80复合物通过“中心旋转”机制及其对不同组蛋白表面的适应性识别,获得了对经典核小体与非经典核小体的双重滑动活性。这种灵活的底物选择性对其在复杂染色质环境中发挥功能至关重要,并为理解染色质动态结构的形成机制提供了新的视角。在活跃转录的基因区域,六聚体核小体的形成往往伴随连接DNA长度的增加,进而可能通过招募INO80参与该区域的核小体定位与分布调控(图4)。
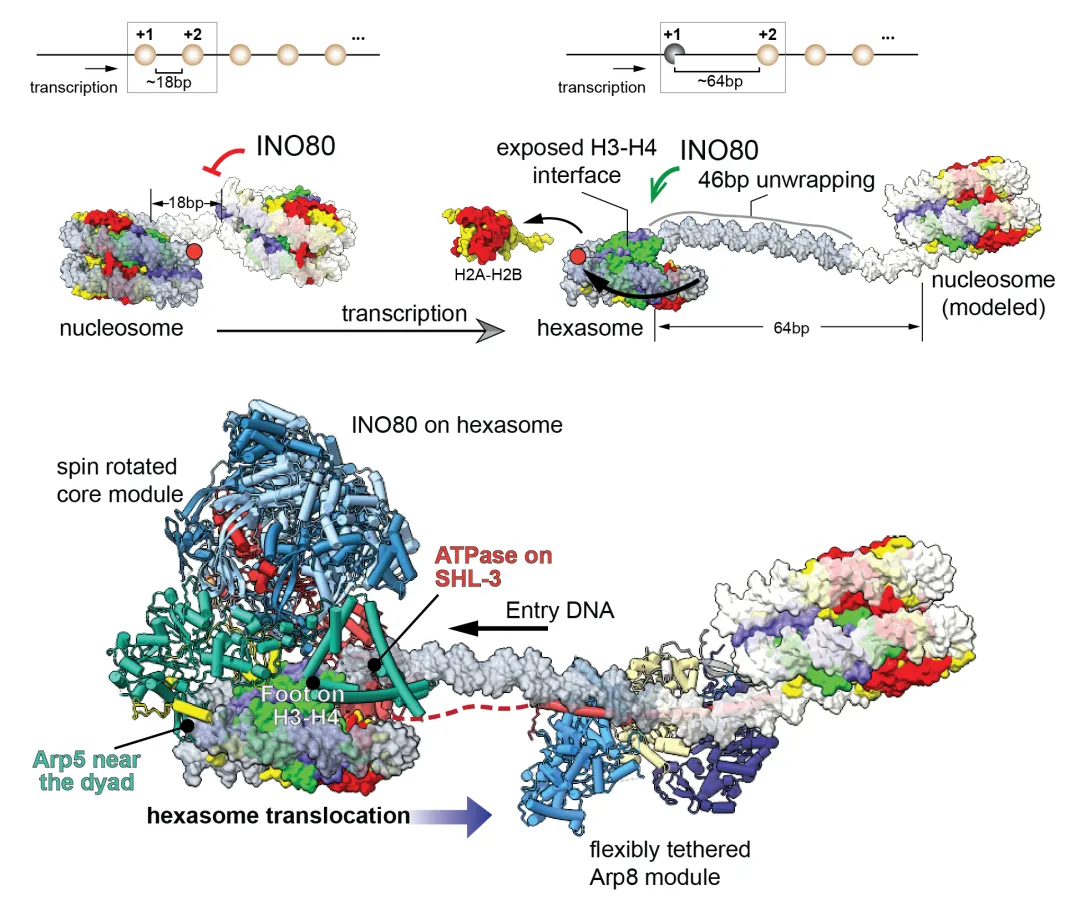 图4 转录延伸过程中六聚体核小体的滑动模式图
图4 转录延伸过程中六聚体核小体的滑动模式图
本研究系统揭示了INO80复合物识别与滑动六聚体核小体的结构基础,为深入探索非经典核小体如何协同组蛋白修饰、组蛋白变体等表观遗传因素参与基因表达调控奠定了重要基础。同时,该研究也为进一步阐明非经典核小体与其他染色质重塑复合物、转录机器及组蛋白伴侣等因子在染色质层面的协同互作机制提供了关键的结构依据。












 沪公网安备31011502400759号
沪公网安备31011502400759号
 营业执照(三证合一)
营业执照(三证合一)


