 Hello,Hello,各位好呀,转眼就来到了六月份,不知道大家收获如何?新的月份,新的开始,祝大家科研顺利、投标必中!
Hello,Hello,各位好呀,转眼就来到了六月份,不知道大家收获如何?新的月份,新的开始,祝大家科研顺利、投标必中!
今天继续为大家分享单细胞多组学研究工具推荐。
本期我们要介绍的是一个能加速单细胞数据处理的工具,rapids-singlecell,利用GPU来加速单细胞转录组数据的处理速度及提升数据的处理规模。
随着AI时代的到来,由于GPU(Graphics Processing Unit)即图形处理器具有高度并行计算能力,其从最初为了图像和视频渲染设计的专用处理器,现在已经广泛用于科学计算、AI、机器学习、数据分析等领域。比如在基因组领域的Parabricks分析工具,Parabricks 通过 GPU 加速常见的二代测序流程,如比对和变异检测,大幅提升分析速度。某些模式下,相较于常规流程,分析时间可以节省十倍以上。对于大规模的NGS下机数据动辄几十个小时的处理时间,是十分有用的。
在单细胞数据处理中,我们之前已经为大家介绍过Seurat V4、Seurat V5、Scanpy等软件分析方法。这些软件作为当前单细胞分析的主流方案,在数据的处理上各有优劣势。另一方面,随着市场上单细胞的单价越来越低,单细胞样本的通量也逐渐上升,同时公共数据库中也存储这大量的数据资源。所以现今分析的细胞数量级也从最初的几万到现在的几十万,甚至百万级别。庞大的细胞量让资源的消耗和运行的时间成指数级增长。就算是以分析大规模数据见长的Scanpy分析起来也很吃力。
那么利用GPU可以加速分析时间以及轻松处理百万级水平的数据么?有解决方案么?答案是有的。 rapids-singlecell 项目旨在将 RAPIDS GPU加速技术应用于大规模单细胞测序数据的分析,替代传统的 CPU 流水线(如 Scanpy、Seurat)中的限速步骤,以面向百万级细胞数据,解决现有工具在大规模数据下的性能瓶颈。
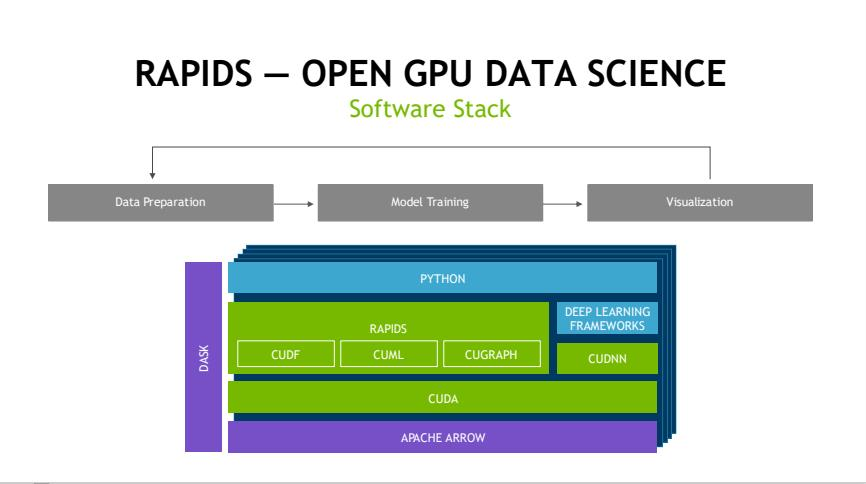
一:RAPIDS生态
现在我们来回想下,在单细胞数据处理中的几个步骤以了解rapids-singlecell 是如何实现加速的:
- 预处理

- 基因表达矩阵加载和转换(cuDF)
- 高变基因筛选
- 数据标准化(如 log-transform)
- 降维

- PCA:通过 PCA 实现 GPU 加速
- t-SNE / UMAP:使用 cuML 对高维数据进行可视化降维
- 邻近图构建

- 使用 GPU 版本的最近邻搜索(cuML)
- 构建邻接图(邻接矩阵)
- 聚类

- 利用 cuGraph 实现 Louvain 和 Leiden 聚类算法
- 对细胞进行图聚类划分
接下来我们进行了一些实操,我们预先写好了一个单细胞的测试脚本,基于Scanpy和rapids-singlecell等工具,覆盖了单细胞转录组分析的前期常见的处理步骤。分别测试了在常规几万个细胞和大规模细胞项目下的表现。
我们定义了几个步骤,分别是:
- 数据的读入(load_data),读入MTX稀疏矩阵文件
- 基础QC部分(basic_qc),在此步我们进行了线粒体等比例的计算,以及相关指标的过滤工作
- 数据预处理步骤(preprocess),在此步我们对数据进行了normalize,Scale等处理,以及高变基因的鉴定、数据的PCA降维、UMAP降维、leiden分群等工作
- Harmony(harmony_integration)去批次,此步为可选步骤,主要是使用harmony进行样本间去批次工作
- UMAP画图(visualize_umap),在此步主要完成了基于分群结果的可视化展示
- 细胞注释(cell_type_annotation),在此步基于了celltypist进行细胞的注释,并且输出相关的注释信息与绘图
- Marker基因分析(marker_gene_analysis),此步进行各亚群的marker基因的鉴定并且输出相关的注释信息与绘图
首先我们测试了在两个样本(~40000 cell)的数据处理效果,我们的测试环境配备了256线程CPU,以及RTX 3090 24G显卡用于rapids-singlecell的计算。同时我们构造了一个装饰器统计每步所用的时间、内存、CPU等消耗.具体运行结果如下所示,通过下图可以很明显的看到每个步骤基本都在10秒左右完成,四万个细胞的处理仅需1分钟就可以完成,并且对于CPU的消耗主要集中在数据读入部分,大概需要2~3个线程,对于内存的消耗主要是在数据的预处理与harmony整合部分,最多占用2G左右的内存。
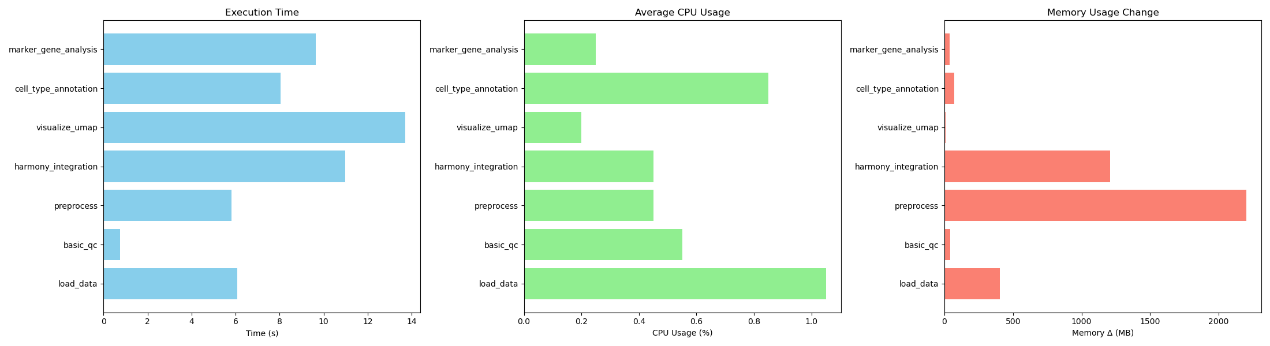
图2:rapids-singlecell等流程在小数据集上的表现
接下来我们来简单查看下数据处理后的结果,可以看到数据的整合,细胞鉴定,marker鉴定的表现还算合理。
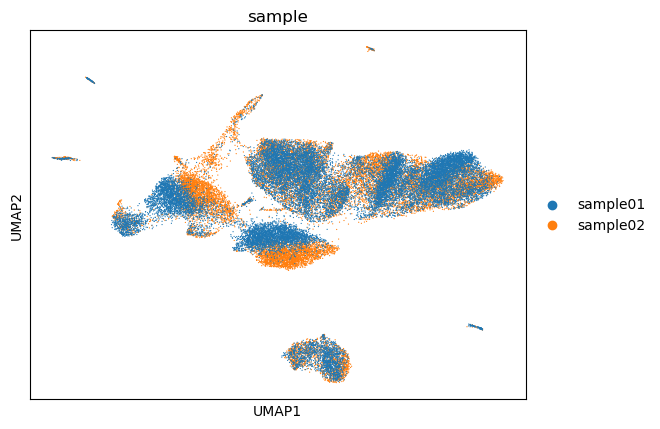
图3 样本去批次前UMAP图
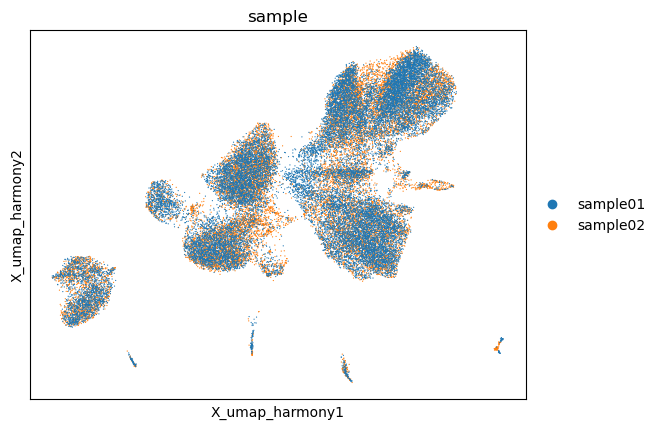
图4 样本去批次后UMAP图
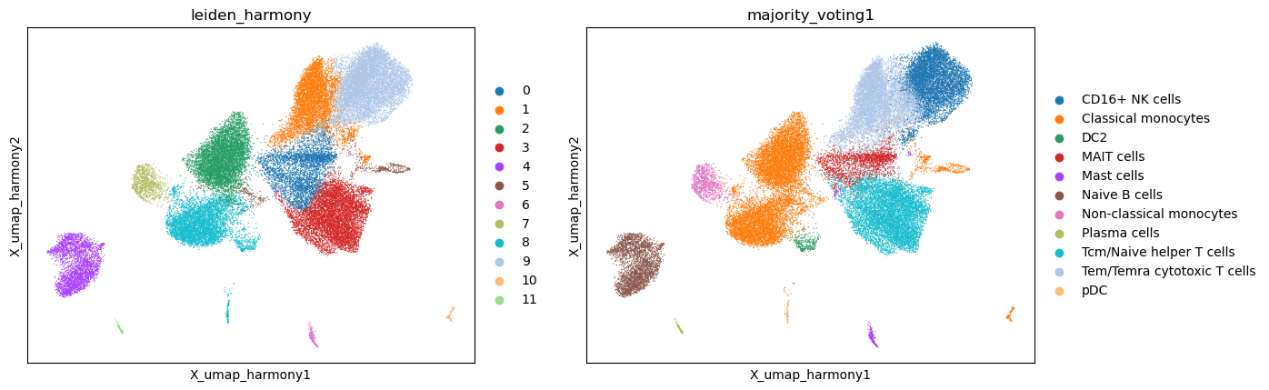
图5 机器注释结果
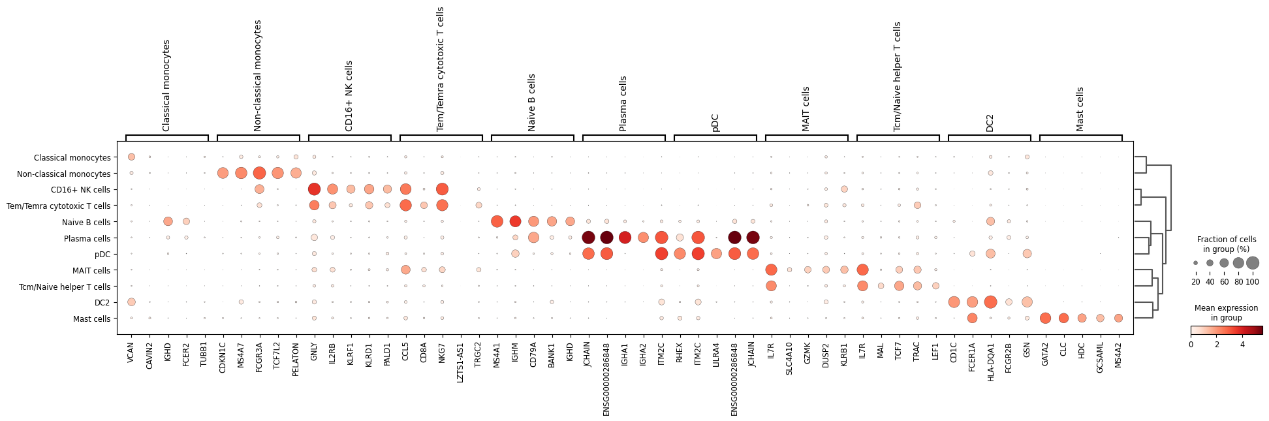 图6 各细胞类型的marker鉴定结果展示
图6 各细胞类型的marker鉴定结果展示
接下来,我们想看下分析流程在更复杂的场景下的表现。我们从GEO数据库中下载了多个肿瘤单细胞数据集,共计1010695个细胞,令我们比较惊讶的是,在GPU仅24G显存的测试环境下,依然可以在半个小时以内完成全部分析。并且这还是在依次读入矩阵的情况下,如果进行多个矩阵同时读入,时间将会进一步压缩。具体的表现如下图所示,可以看到和小型数据集不同的是,数据的读入、预处理、整合成为了耗时的步骤。CPU资源的消耗没有太大的增长,对于内存的消耗最大在数据预处理步骤,大概在40GB左右。
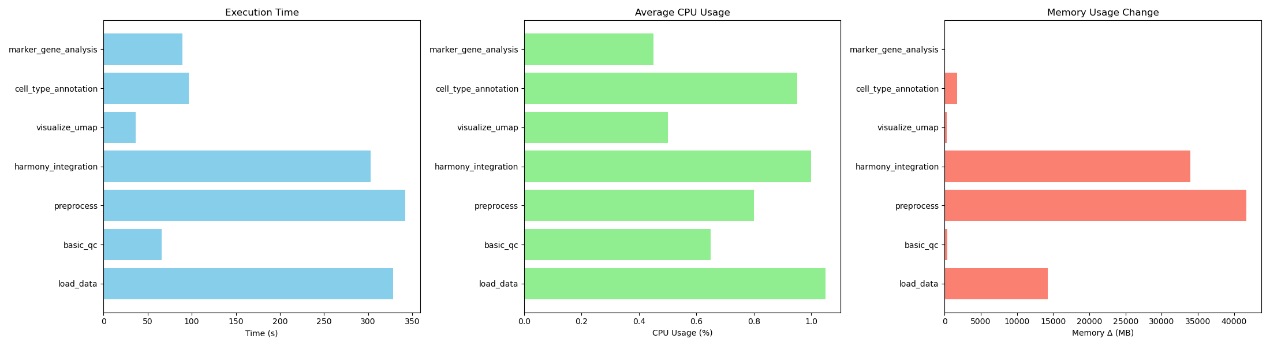
图7:rapids-singlecell等流程在大数据集上的表现
接下来我们来观察下数据的整合后表现,百万级的数据对于整合的挑战是巨大的,基于数据的分群来看,还是可以比较明确的进行区分与降维。基于机器注释的结果,后续细胞注释需要人工进行精细的调整。
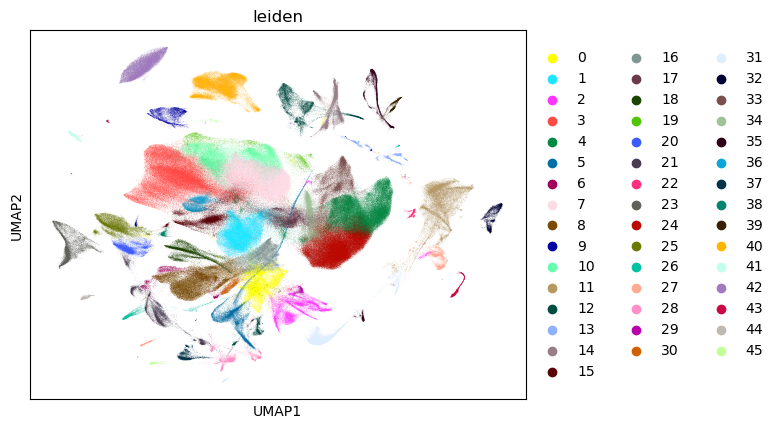
图8:未进行去批次之前的分群情况
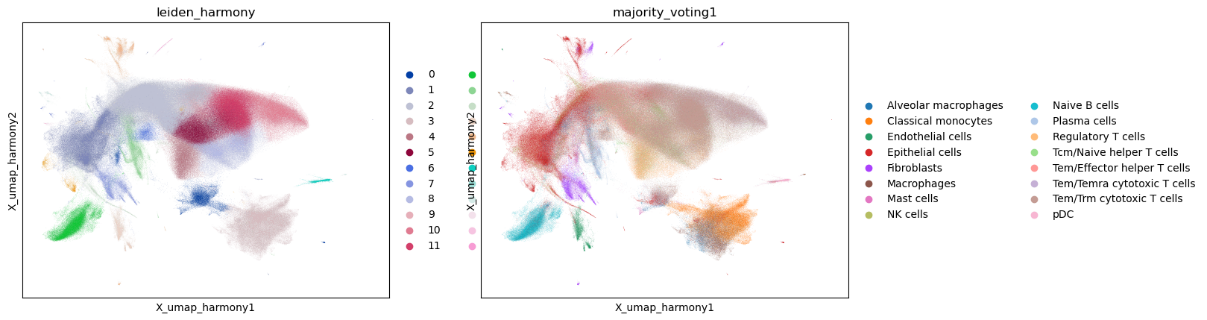
图9:细胞去批次之后的UMAP降维、分群及细胞注释情况
好了,本次测试到这里就结束,大家有任何问题或者后续想看哪些测试也可以留言告诉我们。
乐备实在单细胞多组学研究与软件流程开发中具有丰富的经验,欢迎各位老师同学与我们联系合作。
乐备实是国内专注于提供高质量蛋白检测以及组学分析服务的实验服务专家,自2018年成立以来,乐备实不断寻求突破,公司的服务技术平台已扩展到单细胞测序、空间多组学、流式检测、超敏电化学发光、Luminex多因子检测、抗体芯片、PCR Array、ELISA、Elispot、多色免疫组化等30多个,建立起了一套涵盖基因、蛋白、细胞以及组织水平实验的完整检测体系。
我们可提供从样本运输、储存管理、样本制备、样本检测到检测数据分析的全流程服务。凭借严格的实验室管理流程、标准化实验室操作、原始数据储存体系以及实验项目管理系统,已经为超过3000家客户单位提供服务,年检测样本超过100万,受到了广大客户的信任与支持。











 沪公网安备31011502400759号
沪公网安备31011502400759号
 营业执照(三证合一)
营业执照(三证合一)


