在生物信息学飞速发展的当下,海量组学数据的涌现使传统分析方法面临巨大挑战,而机器学习凭借强大的数据处理与模式识别能力,成为推动该领域发展的核心力量。其中,在肠道微生物组研究中,机器学习的应用尤为突出,为解析微生物与健康的复杂关系提供了全新视角。
人体肠道微生物组:健康与疾病的隐形关联者
人体肠道是一个极其复杂的微生态系统,定植着细菌、古细菌、真菌、微生物真核生物和病毒等多种微生物群落,它们与人类宿主形成紧密的共生关系,共同构成肠道微生物组。据估算,肠道内细菌数量近 100 万亿,其基因组规模约 300 万个基因,是人类宿主基因组的 150 倍。在健康状态下,宿主与微生物群维持动态平衡的生态稳态,这是正常生理功能的重要保障。但受饮食、抗生素、环境及遗传等因素影响,平衡可能被打破,引发生态失调,即微生物群组成的异常改变。大量研究表明,生态失调与炎症性肠病、代谢综合征、心血管疾病、神经退行性疾病及恶性肿瘤等多种疾病密切相关,然而其具体关联机制仍有待深入探索。
组学技术驱动下的肠道微生物组数据洪流
随着对肠道微生物组重要性认识的加深,相关研究显著增加,组学技术的应用为此提供了高通量、高分辨率的数据支撑。宏基因组学技术通过高通量测序,能全面揭示肠道微生物群落的物种组成、基因功能及代谢潜力。16S rRNA 基因测序凭借高特异性和成本效益,广泛用于微生物分类鉴定与多样性分析;全基因组鸟枪测序则可提供更全面的基因组信息,助力深入挖掘微生物功能特征。宏转录组学聚焦微生物群落的整体转录活动,通过 RNA 高通量测序,实时反映特定生理状态下微生物基因的表达变化,为解析动态功能调控提供关键线索。代谢组学对肠道微生物产生的小分子代谢物进行系统检测与定量,直接关联微生物代谢活动与宿主生理病理状态,是连接微生物群落结构与宿主表型的重要桥梁。
机器学习:应对组学数据挑战的核心工具
这些组学技术的联合应用产生了海量多维数据,涵盖微生物分类组成、基因序列、基因表达谱和代谢物浓度等。这些数据规模庞大,且具有高维度、高噪声、非线性及样本异质性强等复杂特征,传统统计分析方法难以应对。例如,处理包含数万甚至数十万微生物特征的数据集时,传统方法常面临维度灾难、特征冗余和计算效率低下等问题,难以挖掘潜在的生物学模式与关联。而机器学习作为人工智能的核心分支,能自动从海量数据中学习潜在规律,构建预测模型或进行特征筛选,有效克服传统方法的局限性,成为推动肠道微生物组研究向纵深发展的关键技术。
机器学习在肠道微生物组研究中的多元应用场景
在肠道微生物组研究中,机器学习方法已广泛应用于多个方向。
疾病诊断与生物标志物发现
在疾病诊断与生物标志物发现领域,监督学习算法作用显著。研究者利用支持向量机、随机森林、逻辑回归和深度学习等算法,以肠道微生物组特征为输入构建疾病预测模型,实现疾病状态的精准分类与预测。如在结直肠癌研究中,通过对患者与健康人群的肠道菌群数据进行机器学习分析,成功筛选出多个具有诊断价值的微生物标志物组合,诊断准确率可与传统临床检测指标媲美。
微生物群落结构解析
无监督学习算法在微生物群落结构分析中表现出色。聚类分析算法能根据微生物群落组成特征,将样本划分为不同亚群,助力识别相似菌群结构的人群亚型,为疾病精准分型与个体化治疗提供依据。主成分分析、t - 分布邻域嵌入等降维算法可将高维微生物组数据映射到低维空间,在保留关键信息的同时实现数据可视化,帮助研究者直观理解群落结构差异与变化规律。
微生物功能预测与网络构建
在微生物功能预测方面,机器学习算法基于已知的微生物基因序列与功能注释信息,构建基因功能预测模型,实现对未知基因功能的高效注释。例如,利用深度学习中的卷积神经网络和循环神经网络分析微生物基因组序列,能显著提高基因预测与功能注释的准确性和效率。此外,整合宏基因组学、宏转录组学和代谢组学等多组学数据,机器学习可构建微生物群落的代谢网络模型,揭示功能协作机制及与宿主代谢的相互作用关系。
关联模式挖掘
在关联分析领域,机器学习算法能有效挖掘微生物组特征与环境因素、宿主表型之间的复杂关联。通过构建回归模型,可量化饮食成分、生活方式等环境因素对肠道菌群结构的影响程度;利用关联规则挖掘算法,能发现与特定疾病表型相关的微生物组合模式,为揭示疾病发病机制提供新思路。
机器学习应用中的挑战与应对策略
尽管机器学习在肠道微生物组研究中成果显著,但仍面临重大挑战。监督学习模型依赖训练数据的数量和质量,而样本量小、标签分布不成比例、实验方案不一致或元数据缺失等问题,可能导致模型缺乏可重复性。如两项结直肠癌患者肠道菌群的荟萃分析发现,虽均观察到生态失调,但特定人群特有的细菌多样性在其他研究中未出现。
为解决相关问题,研究者正积极探索方法。创建人类肠道微生物群数据存储库是重要举措,结合数据共享透明度的提高,研究者可对各种研究进行荟萃分析,确定针对某些疾病的稳健生物标志物或生态失调指标。许多研究者认为,存储库中预处理数据的可用性能最大限度减少技术偏差并降低计算成本。同时,改进数据披露准则和推广更易于访问的框架,也有助于开发更准确可靠的机器学习模型。制定统一的数据采集、处理与报告标准,要求研究者公开原始数据、实验方法与分析流程,可提高研究透明度与可追溯性。推广标准化实验操作流程,能减少技术偏差对数据质量的影响,为机器学习模型提供高质量训练数据。
开发易用高效的分析框架是降低技术门槛、促进机器学习普及应用的关键。针对肠道微生物组数据特点,研发集成化数据分析工具与平台,实现数据预处理、特征筛选、模型构建与结果可视化的一站式分析,可帮助研究者更便捷地应用机器学习方法,提高数据分析效率,减少因分析方法差异导致的结果不一致问题。
未来展望:从关联观察到临床转化的跨越
机器学习在分析肠道微生物群多组学研究产生的海量数据中发挥关键作用,推动了微生物与疾病新联系的发现。随着组学分析数据可用性的增加,数据存储库、指南和分析框架的完善,正促进该领域从观察关联研究向实验因果推断和临床干预转变。未来,随着技术的持续创新与跨学科合作的深入,机器学习在生物信息学领域的应用将更加广泛,为揭示生命奥秘、推动疾病防治提供更强大的技术支撑。
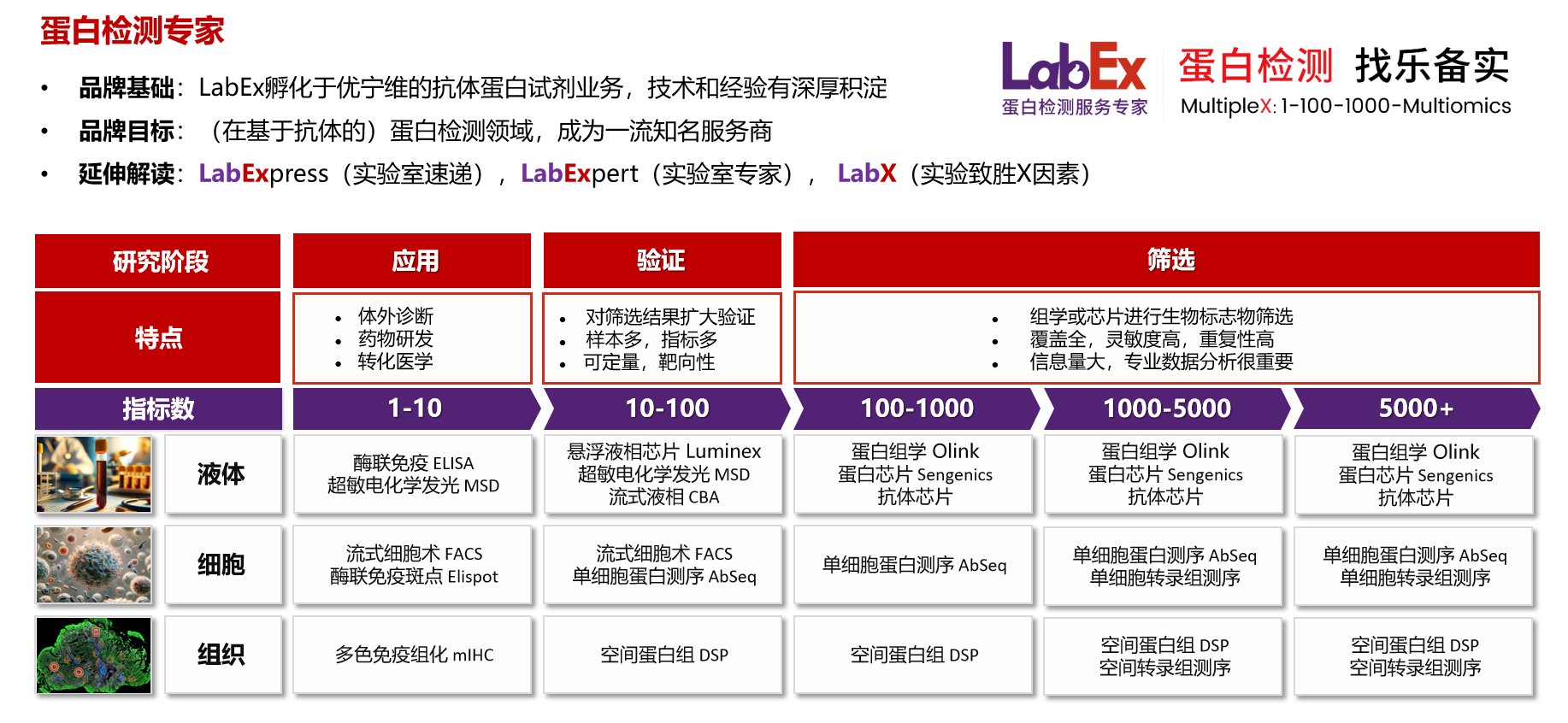
乐备实是国内专注于提供高质量蛋白检测以及组学分析服务的实验服务专家,自2018年成立以来,乐备实不断寻求突破,公司的服务技术平台已扩展到单细胞测序、空间多组学、流式检测、超敏电化学发光、Luminex多因子检测、抗体芯片、PCR Array、ELISA、Elispot、多色免疫组化等30多个,建立起了一套涵盖基因、蛋白、细胞以及组织水平实验的完整检测体系。
我们可提供从样本运输、储存管理、样本制备、样本检测到检测数据分析的全流程服务。凭借严格的实验室管理流程、标准化实验室操作、原始数据储存体系以及实验项目管理系统,已经为超过3000家客户单位提供服务,年检测样本超过100万,受到了广大客户的信任与支持。











 沪公网安备31011502400759号
沪公网安备31011502400759号
 营业执照(三证合一)
营业执照(三证合一)


