引言
人类心脏发生是一个高度协调的发育过程,涉及多个细胞阶段的逐步分化,包括人类胚胎干细胞(hESC)、心脏中胚层(MES)、心脏前体细胞(CP)和成熟心肌细胞(CM)。近年来的研究表明,染色质三维结构的动态变化在基因表达的时空调控中发挥关键作用。Hi-C技术为在全基因组范围内揭示染色质空间组织提供了有力工具,而整合多组学数据进一步深化了我们对心脏发育中结构-功能关系的理解。心脏发育中染色质动态调控网络:在人类心脏发生过程中,基因组的三维结构经历着剧烈而精确的重编程。这些动态变化,包括区室转换和染色质可及性的改变,构成了一个高级的调控网络,协同激活心脏特异性基因表达程序,并沉默多能性基因,最终驱动细胞命运向心肌细胞定向分化。本文系统总结了染色质构象变化、区室化动态、拓扑关联结构域(TAD)重组以及染色质可及性在心脏发生过程中的作用,并探讨了其与基因表达调控的关联。

一、心脏发生中染色质区室化的动态特征
(一)A/B区室整体稳定性与局部动态性
在500 kb分辨率下对Hi-C数据进行分析发现,人类心脏发生各时期细胞中基因组整体呈现A和B两种区室各占一半的格局。A区室通常对应常染色质,富含活跃转录基因;B区室则与异染色质相关,基因表达较低。研究显示,在整个分化过程中,区室整体结构保持较高稳定性,仅约19%的基因组区域发生了区室转换,其中以单向转换(B→A或A→B)为主,回复性转换(A→B→A或B→A→B)占比较低。

(二)区室互作模式与染色质压缩状态
研究进一步发现,位于相同区室类型(A-A或B-B)内的染色质区域之间具有更强的顺式互作。在分化过程中,最强顺式互作由A区室逐渐转向B区室,导致B-B区室中大于10 Mb的远距离互作显著增加。相比之下,A-A和B-B区室间的反式互作在不同分化时期相对保守。这些现象提示,随着分化进行,异染色质区域可能经历进一步压缩,从而增强其长距离相互作用。

二、三维基因组结构与基因表达的相关性
(一)区室转换与差异基因表达
通过整合RNA-seq数据,研究发现差异表达基因具有明显的细胞类型特异性。在CP和CM中上调的基因显著富集于从B向A转换的基因组区域。基因本体(GO)分析显示,CM中高表达基因主要参与心脏发育及代谢相关通路。这一结果说明,许多心脏发育关键基因的表达激活可能依赖于其所在染色质区域由异染色质向常染色质的转换。
值得注意的是,尽管回复性转换区域仅占全基因组的4%,但其在CP时期短暂转换为A区室时,内部基因(如BMPER和CXCR4)表达水平显著上升;相反,中胚层调节因子EOMES在其所在区域转换为B区室时表达下调。这表明区室状态的短期动态变化可直接调控基因表达水平。

(二)TAD动态与区室化的相对独立性
利用Directionality Index和Insulation Score两种方法评估TAD边界在分化中的变化,发现大多数TAD边界在不同时期保持稳定。值得注意的是,A区室中TAD的平均尺寸显著小于B区室。从hESC向CM分化过程中,TAD边界消失的区域主要富集于组成型B区室或由A转换为B的区域,这与异染色质逐渐压缩的现象一致,提示部分TAD可能在分化中与邻近区域发生融合。
然而,TAD边界的变化与基因表达并未显示直接相关性。新形成的TAD边界更倾向于与邻近基因的激活相关,但与区室转换无显著关联。这表明TAD的动态调整在一定程度上独立于A/B区室化,可能通过其他机制参与基因表达调控。
三、染色质可及性与三维基因组组织的关联
(一)ATAC-seq peaks的区室特异性分布
染色质可及性分析显示,在分化过程中,A区室中ATAC-seq信号显著增加,而B区室中则减少。CM特异的开放区域主要富集在B→A转换区域,其基序分析识别出GATA、NKX等心脏发育关键转录因子结合位点。
(二)转录因子结合与可及性动态
通过整合公共ChIP-seq数据,研究发现转录因子GATA4、NKX2-5和TBX5在CM时期的结合位点特异性富集于CM特异的开放区域。值得注意的是,GATA4在hESC中的结合位点并未富集于时期特异性开放区域,而是广泛分布于组成型开放区域中,提示GATA4可能在早期发挥“看家”功能。进一步比较显示,GATA4在CP和CM中的富集程度相似,而NKX2-5和TBX5更显著富集于CM特异性区域,表明GATA4可能是心脏分化的早期驱动因子,NKX2-5和TBX5则协同强化下游调控网络。

四、三维基因组结构调控特定基因表达的机制
(一)TTN基因的顺式调控机制
肌联蛋白基因TTN是人类最大的蛋白编码基因,其突变与家族性扩张型心肌病密切相关。研究显示,在心脏发生过程中,TTN所在区域从B区室转换为A区室,其两个转录起始位点(TSS)的染色质可及性显著增加,表达水平明显上调。这两个TSS分别调控全长转录本和 shorter Cronos TTN异构体,均在心脏发育中发挥重要作用。
TTN的开放区域包含GATA4、NKX2-5、TBX等心脏关键转录因子的结合位点,且与CTCF结合位点重叠。值得注意的是,位于TTN转录终止位点(TTS)的CTCF motif方向与基因内部位点相反,提示可能形成基因内环结构。在分化过程中,CTCF占据减少,TTN的TSS与基因内部互作减弱,而与上游区域互作保持稳定,表明在hESC中,CTCF可能通过介导基因内环抑制TTN表达,而分化中CTCF占位减少则可能解除这一抑制,促进表达激活。
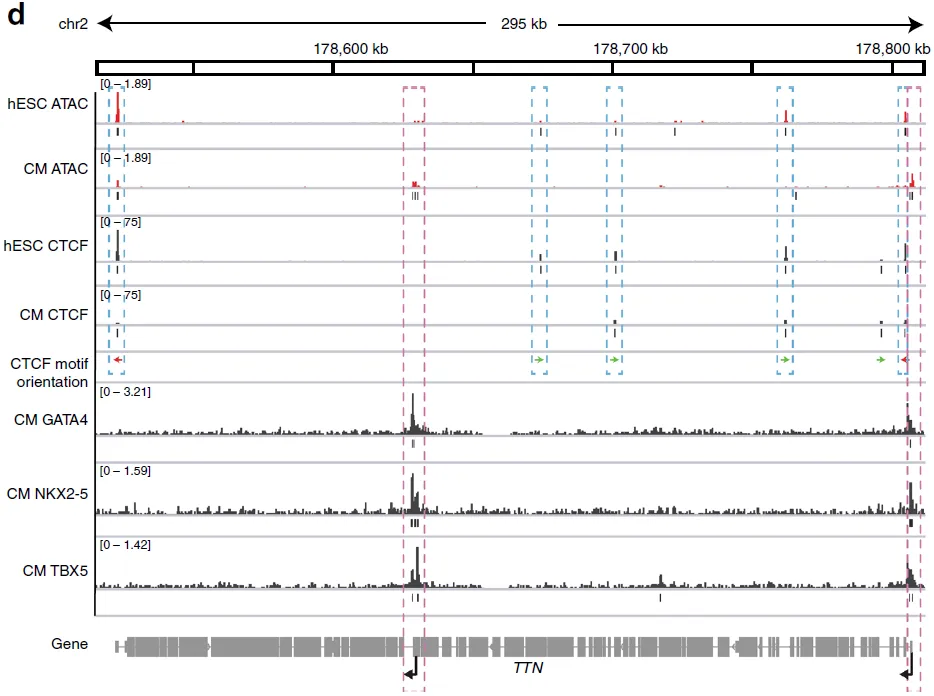
(二)TTN的反式互作与RBM20介导的剪切工厂模型
除顺式调控外,TTN还与多个基因组位点存在反式互作,包括心脏基因CACNA1C和CAMK2D。这些基因均受RNA结合蛋白RBM20的调控,该蛋白通过促进外显子互斥参与心脏特异性异构体生成,其突变同样与心肌病相关。研究进一步发现,在CM细胞中,16个RBM20调控的可变剪切基因均表现出特异性反式互作增强。FISH实验证实,与hESC相比,TTN与CACNA1C等位点在CM中的空间距离显著缩短。这些位点同时从核边缘向核中心移动,与其从B到A的区室转换一致,提示三维基因组重排可能促进其共定位与表达激活。
功能实验表明,转录抑制剂放线菌素D处理可显著减弱TTN与CACNA1C、CAMK2D的空间邻近性,说明该互作依赖于转录过程。通过CRISPR/Cas9敲除TTN启动子(影响mRNA生成)而非编码区突变(不影响转录)会导致RBM20蛋白聚集减弱,表明TTN pre-mRNA对招募RBM20至关重要。最终,RBM20敲除导致TTN、CACNA1C和CAMK2D异常异构体产生,并破坏其空间互作,证实RBM20通过介导反式互作形成“剪切工厂”,协调多基因的可变剪切过程。

五、总结与展望
本研究系统揭示了人类心脏发生中三维基因组的动态变化及其与基因表达调控的密切关系。区室转换、TAD重组和染色质可及性变化共同构成了一个多层次调控网络,控制心脏发育关键基因的时空表达。特别值得注意的是,TTN及其他RBM20靶基因的反式互作机制为理解高级染色质结构在RNA加工中的作用提供了新视角。多组学技术解析三维基因组与功能调控: 未来,结合Hi-C、ATAC-seq、ChIP-seq和RNA-seq等多组学技术,将能更精细地绘制染色质结构动态图谱,并直接关联基因调控输出。这种整合性研究策略将是彻底解密发育、疾病乃至进化中基因表达调控核心机制的关键。未来研究需进一步整合多组学与单细胞技术,深入解析心脏发育与疾病中三维基因组调控的精确分子机制。
乐备实是国内专注于提供高质量蛋白检测以及组学分析服务的实验服务专家,自2018年成立以来,乐备实不断寻求突破,公司的服务技术平台已扩展到单细胞测序、空间多组学、流式检测、超敏电化学发光、Luminex多因子检测、抗体芯片、PCR Array、ELISA、Elispot、PLA蛋白互作、多色免疫组化、DSP空间多组学等30多个,建立起了一套涵盖基因、蛋白、细胞以及组织水平实验的完整检测体系。
我们可提供从样本运输、储存管理、样本制备、样本检测到检测数据分析的全流程服务。凭借严格的实验室管理流程、标准化实验室操作、原始数据储存体系以及实验项目管理系统,已经为超过3000家客户单位提供服务,年检测样本超过100万,受到了广大客户的信任与支持。











 沪公网安备31011502400759号
沪公网安备31011502400759号
 营业执照(三证合一)
营业执照(三证合一)


